In questo articolo vediamo le strategie per incrementare le performance del nostro database, fermo restando che tali performance dipendono da molti fattori, in primis l'utilizzo che si fa del database e le operazioni realmente critiche utilizzate dalla nostra applicazione. Ci concentreremo su due aspetti:
- parametri di configurazione che hanno effetto sulle prestazioni globali del database;
- profilazione di query specifiche, al fine di ottimizzare quelle particolarmente lente.
Non ci occuperemo dell'ottimizzazione dei cluster, in quanto materia piuttosto complessa, limitandoci a rimandere alle risorse reperibili in rete.
Configurazione del sistema
Un parametro molto importante da tenere in considerazione è la memoria riservata. Considerando che anche in un server dedicato a Neo4j, un porzione della memoria totale viene utilizzata dal sistema operativo, maggiore è la quantità di memoria riservata, migliori sono le performance globali del server.
Ma attenzione: se la memoria poi effettivamente disponibile sul server è minore di quella riservata, il sistema operativo effettuarà molte operazioni di swap su disco, con un impatto molto negativo sulle prestazioni.
Poiché Neo4j è eseguito sulla Java Virtual Machine, la quantità massima di memoria utilizzata dal processo si può impostare con l'opzione -Xmx, oppure utilizzando il parametro dbms.memory.heap.max_size. Ad esempio, per impostare la massima memoria utilizzabile a 8 GB, si può scrivere, nel file di configurazione neo4j.conf:
dbms.memory.heap.max_size=8GIl tuning della memoria passa anche per la definizione della dimensione della cache. La cache è molto importante: maggiore è la porzione di grafo che si trova in memoria, migliori sono le performance globali delle query di ricerca. Se non specificato diversamente, Neo4j alloca il 50% della memoria disponibile per la cache. Possiamo giocare su questo parametro per ottenere prestazioni diverse a seconda del nostro caso d'uso. Ad esempio, per impostare un dimensione di 3GB, possiamo indicare, ancora sul file di configurazione:
dbms.pagecache.memory=3GTramite il file di configurazione si possono modificare molti parametri, elencati in modo esaustivo nella documentazione ufficiale. Segnaliamo di seguito i più importanti:
- dbms.threads.worker_count: numero di thread. Aumentare questo numero può essere utile in caso di applicazioni in cui sono presenti molte richieste concorrenti;
- cypher.statistics_divergence_threshold: valore numerico (da 0.0 a 1.0, per default 0.75) che modifica il comportamento del planner delle query Cypher. Un valore più basso comporta un planner più reattivo, utile in caso di condizioni di database e utilizzi più "stabilizzati" nel tempo;
- metrics.*: vari parametri per abilitare la generazione di vari tipi di metriche relative al Garbage Collector, all'utilizzo della memoria, della rete, della cache, etc...
Profilazione delle query e piani d'esecuzione
Come database consideriamo un altro esempio tratto da GraphGist: il pathway metabolico, l'insieme delle reazioni chimiche coinvolte nei processi metabolici delle cellule. Consideriamo la query n. 4 del grafo in questione, quella che riguarda la ricerca degli enzimi che catalizzano la produzione di NADH:
MATCH (e)-[:CATALYSES]->()-[:PRODUCES]->(m {name:'NADH'}) RETURN e.namePartendo da un nodo qualsiasi, questa query cerca i pattern che, passando per una relazione CATALYSES e una PRODUCES, giungono al nodo NADH.
Cypher dà la possibilità, tramite l'istruzione EXPLAIN, di verificare il piano di esecuzione della query, semplicemente anteponendolo alla query. L'altra istruzione da ricordare è PROFILE, che oltre a mostrare il piano di esecuzione, esegue la query e ne profila le operazioni. Evidentemente, la più affidabili tra queste due istruzione è l'ultima, sebbene non sempre possiamo lanciare la profilazione perché è molto onerosa (soprattutto se siamo in produzione con molti dati).
PROFILE
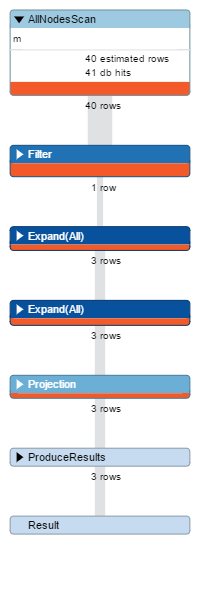
MATCH (e)-[:CATALYSES]->()-[:PRODUCES]->(m {name:'NADH'}) RETURN e.nameIl risultato è illustrato in figura:
Intanto notiamo che il planner ha invertito la ricerca, partendo dal nodo m invece che dal nodo e, reputando giustamente più conveniente filtrare prima, e quindi cercare le relazioni. La prima operazione eseguita è AllNodesScan, ossia la scansione di tutti i nodi del database. In questo caso le entità coinvolte erano solo 40, ma immaginiamo un database in produzione con migliaia di nodi: le performance avrebbero avuto rapidamente un degrado.
Un primo passo è quello di facilitare ulteriormente il lavoro al planner, indicando ad esempio la Label del nodo NADH, cambiando la query in:
PROFILE
MATCH (e)-[:CATALYSES]->()-[:PRODUCES]->(m:Molecule {name:'NADH'}) RETURN e.nameSe la lanciamo, stavolta avremo un NodeByLabelScan ossia la scansione di tutti i nodi con una certa Label. Ora le righe coinvolte sono 22. Nel nostro caso di esempio è un miglioramento di circa il 50%. Vediamo, infatti, che il numero totale di DbHits (numero di operazioni elementari eseguite dal motore di storage) è passato da 94 a 58.
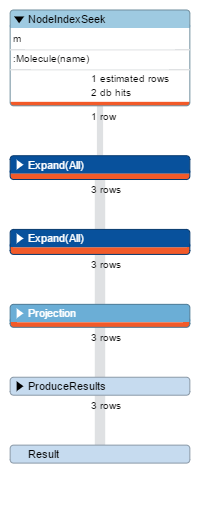
Un ulteriore miglioramento può essere effettuato ricorrendo all'indicizzazione, che vedremo in dettaglio nella prossima lezione. Indicizziamo il campo Name sulla Label Molecule:
CREATE INDEX ON :Molecule(name)Se ora rilanciamo la profilatura, otteniamo il piano rappresentato nella figura seguente:
Vediamo che la prima operazione eseguita stavolta è un NodeIndexSeek, ossia una scansione tramite indice che ha coinvolto una sola riga e 2 sole operazioni elementari. Il numero totale di DbHits è sceso a 15.
Se vuoi aggiornamenti su Database inserisci la tua email nel box qui sotto: